강의 및 실습 스케쥴
8월 4일(월)
9:00~12:00 성신여대 김상태 (A235 실험실)
- DNA 추출 이론 강의
13:00~16:00 성신여대 김상태 (A235 실험실)
- High-Molecular-Weight DNA 추출 1일차
8월 5일(화)
9:00~12:00 성신여대 김상태 (A235 실험실)
- High-Molecular-Weight DNA 추출 2일차
- DNA 전기영동, Qubit에
의한 정량, 정성 분석 의뢰
- Nanopore 이론 강의
13:00-16:00 성신여대 최상철 (A235 실험실)
- Organelle DNA assembly 이론 및 실습
8월18일(월)
9:00~16:00 ONT 안종화, ㈜필코리아 한종민 (A235 실험실)
- Library작성 실습
- Nanopore sequencing running
8월 25일(월)
9:00-12:00 ONT 안종화, ㈜필코리아 한종민 (A235 실험실)
- 결과 생성 및 sequencing 결과
분석
13:00-16:00 성신여대 최상철 (B-109 PC실)
- Long-read에 의한Mitochondrial genome assembly
- Short-read에 의한 polishing
본 워크숍은 Oxford Nanopore Technologies (ONT)로부터
600만원 상당의 물품 후원과 ㈜LAS의 110만원 상당의 MGI short-read sequencing service 후원으로
진행됨을 알려드립니다.
교육자료
1. (PPT) 1 일차 강의 Introduction, HMW DNA 추출 (김상태)
2. (워크숍관련논문) High-Molecular-Weight DNA 추출 논문 및 protocol: Kang et al 2023. APPS
3. (참고논문) CTAB DNA 추출 original 논문: Doyle and Doyle 1987, modified by Sangtae Kim
4. (자료) Filter binding method에 의한 상업용 kit protocol
5. (참고논문) FTA card에 의한 DNC 추출, 보존 및 sequencing 논문: Kim et al. 2016. KJNC
6. (참고논문) Biological nomenclatural system의 안정성에 대한 논문: Jimenez-Mejias et al (2024)
7. (PPT) Nanopore data를 이용 organelle genome assembly (최상철)
8. (워크숍관련논문) POLAP: 나노포어데이터에 특화된 세포소기관 유전체 조립 pipeline 논문: Choi and Kim 2025. JPB
9. (자료) Ligation sequencing gDNA barcoding kit ONT
14. (PPT) 4일차 강의 POLAP running 두번째 자료
14. (참고논문) 참취 충영 발생논문 Boo et al. (2025) Plant, Cell & Enviornment 한빛사 link

HMW DNA 추출 결과
|
# |
ID |
Sci. Name |
Kor. Name |
Qubit (ng/ul) |
Nanopore OD260/280 |
Nanopore OD260/230 |
|
1 |
C_che |
Crepidiastrum chelidoniifolium |
까치고들빼기 |
200 |
1.9 |
1.7 |
|
2 |
L_acr |
Lysimachia acroadenia |
섬까치수염 |
560 |
1.85 |
1.97 |
|
3 |
V_car |
Viburnum carlesii |
분꽃나무 |
56.8 |
1.86 |
1.77 |
|
4 |
P_cuc |
Ponerorchis cucullata |
구름병아리난초 |
70.8 |
1.9 |
1.54 |
|
5 |
H_hon |
Hemerocallis hongdoensis |
홍도원추리 |
206 |
1.84 |
1.85 |
|
6 |
D_pan |
Deutzia paniculata |
꼬리말발도리 |
314 |
1.84 |
1.99 |
|
7 |
V_kap |
Viola kapsanensis |
갑산제비꽃 |
126 |
1.85 |
1.91 |
- 모든 시료는 Kang et al. (2022)에서 nuclei extraction step을 제외한 방법으로 실시한 결과이고, 3, 4번 시료는 결과의 DNA 농도와 260/230의 수치가 높아 AMPure XP (Beckman Coulter, Brea CA, USA) bead 정제법에 의해 다시 한번 정제한 결과임.
- 7번 시료는 끈적이는 carbohydrate가 너무 많아 Kang et al. (2022)의 protocol에서 buffer를 2배로, 시료는 1/2로 하여 시도해 보았지만, 여전히 추출된 DNA가 너무 적었음. 그래서 본 실험의 목표가 mitochondrial genome 이니, HMW DNA 추출방법을 포기하고, 기존의 kit에 의한 방법을 이용하기로 결정하였고, 이 경우 일반적으로 약 10kbp에서 DNA length peak를 보이지만, 이 길이로도 mt genome assembly에는 전혀 지장이 없다고 판단하였기 때문임. 기존의 commercial kit들 중에 Intron 사의 DNA 추출킷트가 carbohydrate에 대한 영향이 가장 적다고 알려져 있어 이를 이용해 추출하였고, 마찬가지로 AMPure XP (Beckman Coulter, Brea CA, USA) bead에 의한 정제를 다시 실시하여 보다 순수하게 하고, 또한 농축 시켰음. 그 결과 Nanopore 실험에 적절한 시료를 확보할 수 있었음.
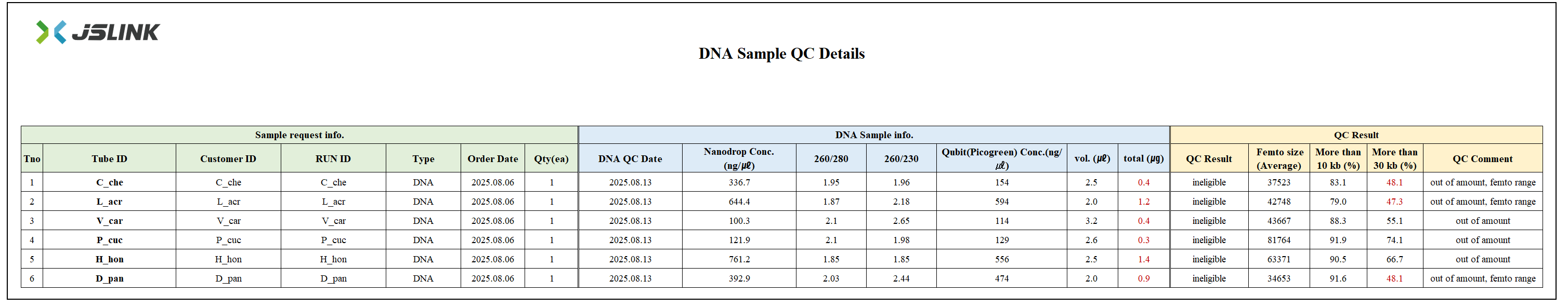
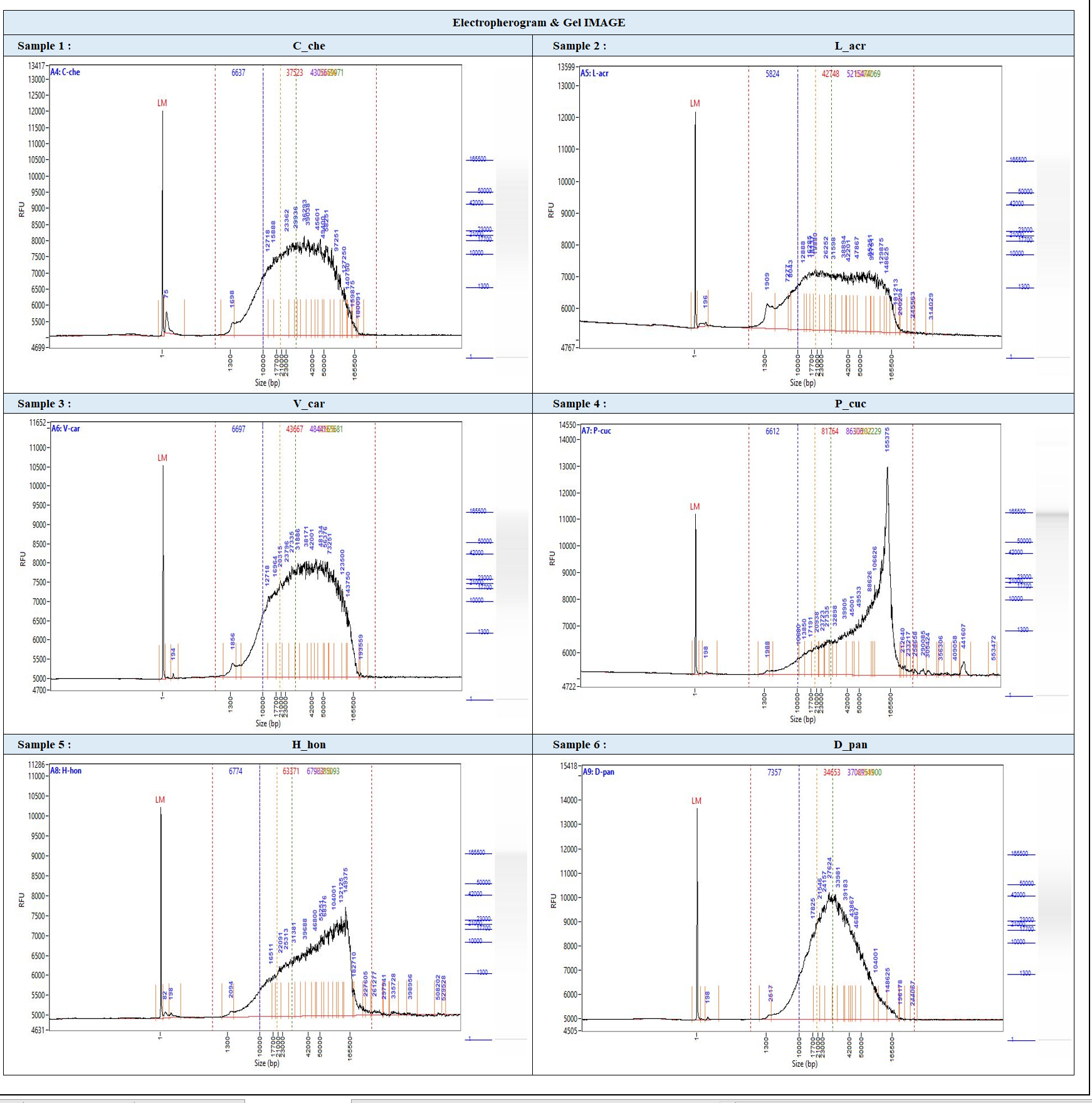
Nanopore sequencing을 위한 QC 결과: JSLINK (https://jslink.co.kr/about)에 의뢰하여 Femto Pulse 로 측정함.
- 4조와 5조의 DNA가 가장 좋은 High-Molecular-Weight DNA 추출 결과를 보임. 둘 다 peak 가 150 kbp 정도이며, 30 kbp 이상되는 fragment의 %가 각각 74%, 67%에 달함.
Nanopore sequencing 결과:
1) First RUN (8월 18일 워크숍 당일) --- Balanced sequencing ON, Old library kit 사용(6개월 정도 보관됨)
123조 Nanopore report - 1st run 456조 Nanopore report - 1st run
- 1,2,3조는 MinIon으로, 4,5,6조는 GridIon으로 실행.
- 실험 시작 30분 후, MinIon은 다소 짧은 서열이 생성되는 것을 확인했지만, GridIon은 N50가 12 kbp 정도의 정상적인 시퀀싱이 이루어짐.
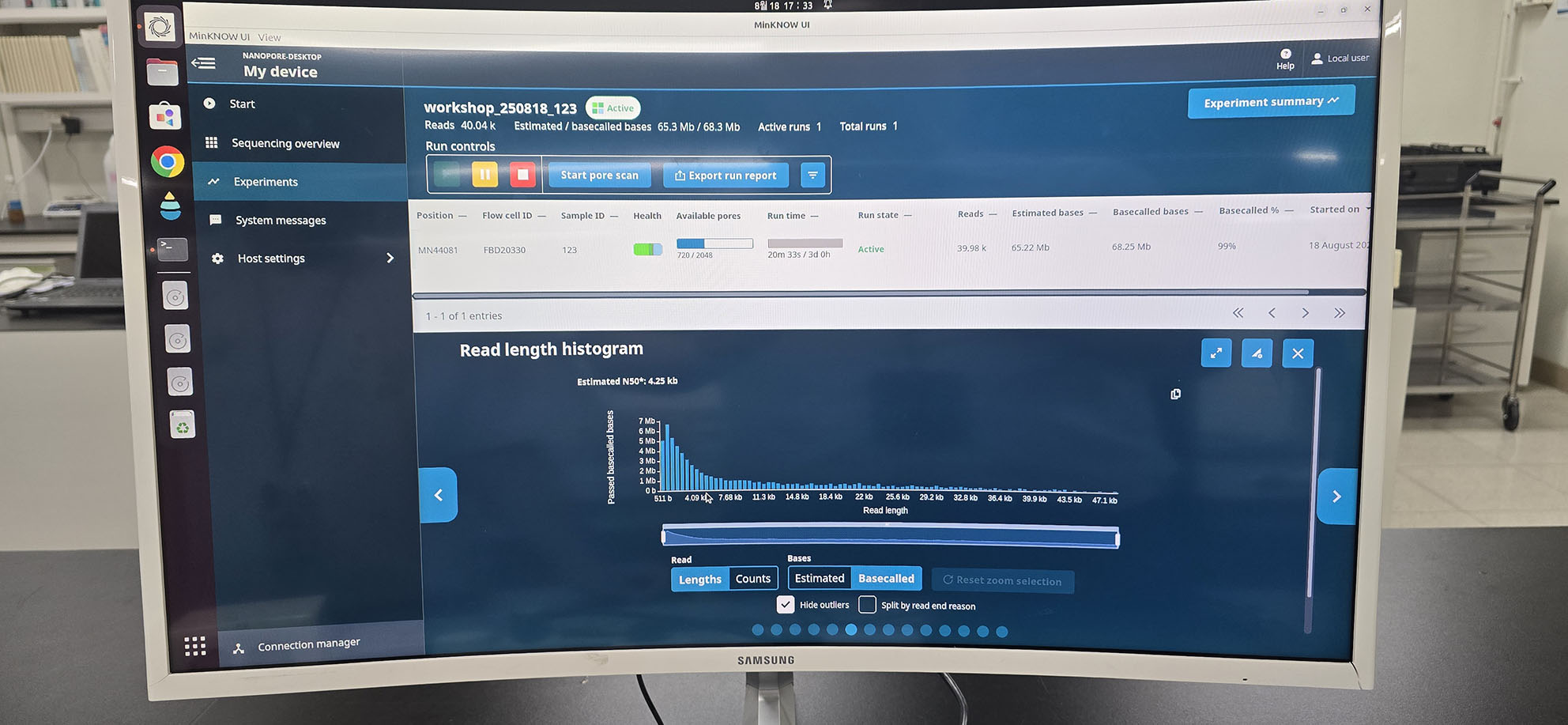
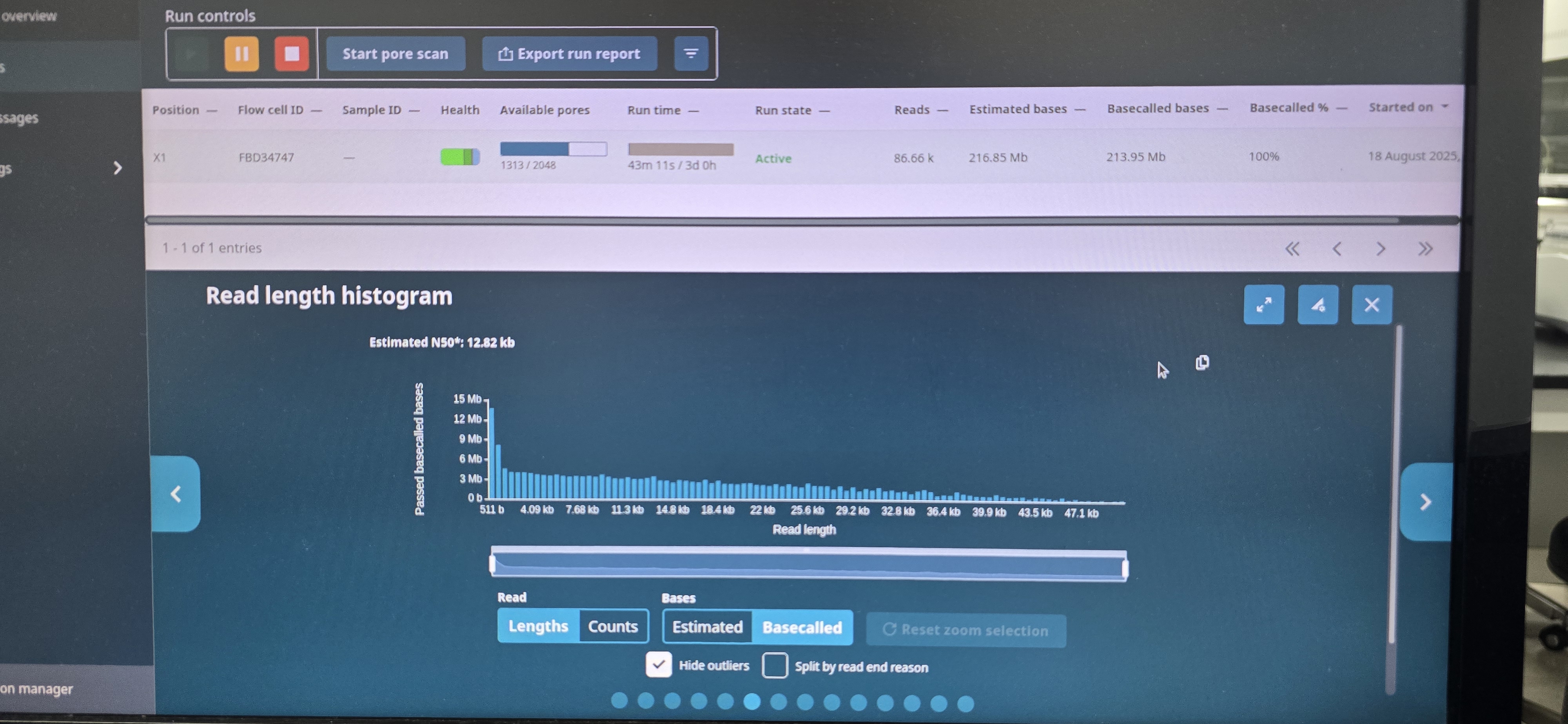
<MinIon> <GridIon>
- 하지만 약 14시간 경과 후 두 실험 모두 N50가 400 bp정도 되는 매우 이상한 결과가 나옴.
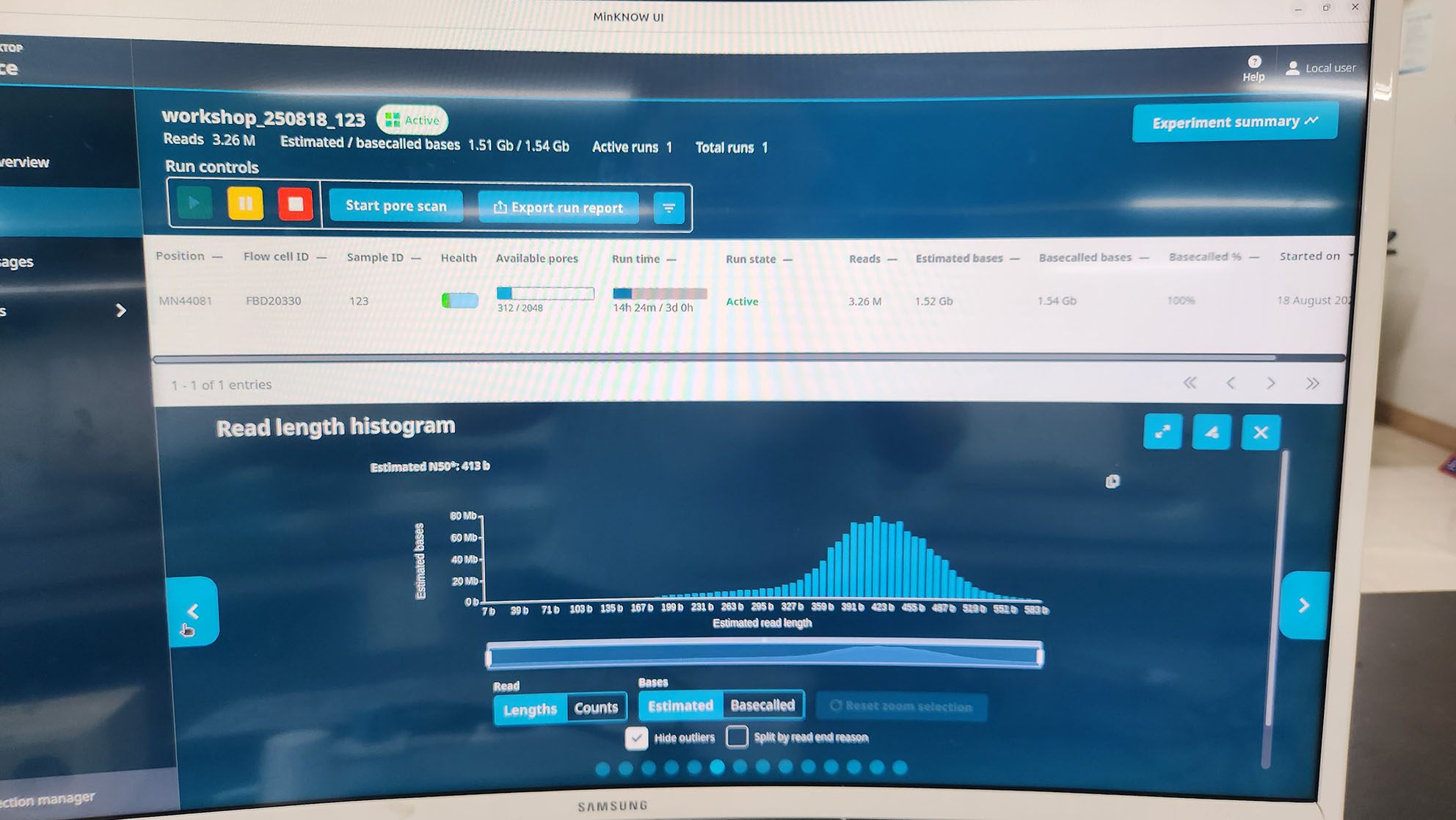
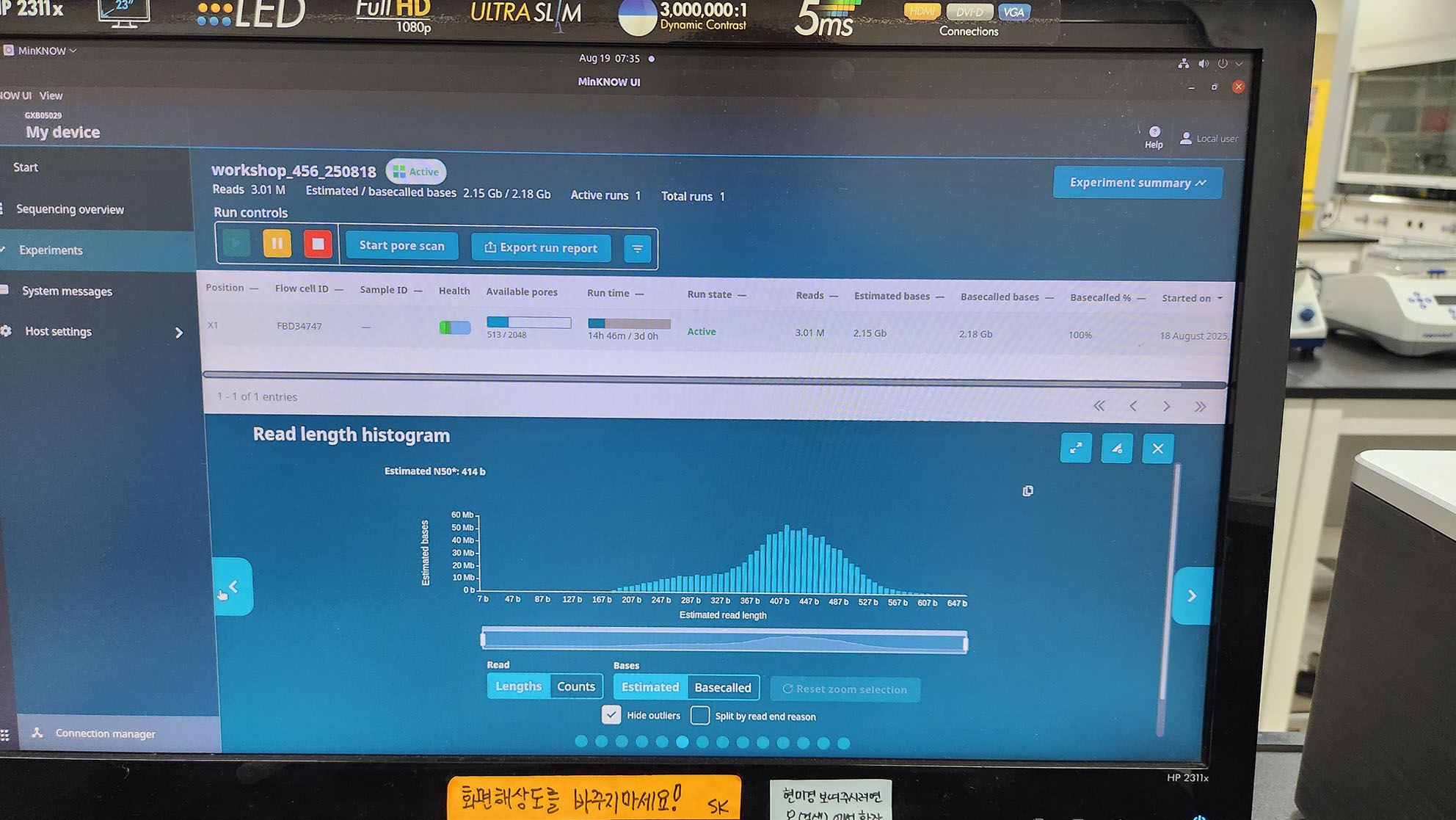
<MinIon> <GridIon>
2) Second RUN --- Balanced sequencing OFF, New library kit 사용 (조교들이 수행함)
- GridIon과 MinIon 실험을 멈추고 flowcell을 washing 후 pore 갯수는 각각 약 250개와 100개만 남아 매우 나쁜 상태였음.
- First Run에서의 실패 원인이 될 수 있는 것이 6개월된 old kit 사용일 수 있어 new library kit를 사용하였고,
소프트웨어가 새로이 업데이트된 Balanced sequencing mode가 문제인 것으로 생각되어 이를 off 함.
- 250개 pore 가 남은 flowcell을 이용하여 6조의 시료 1개 만을 사용하여 running 함.
- First Run에서 얻은 결과를 분석해 본 결과 그나마 6조의 것이 가장 5 kbp 이상의 read 갯수가 가장 많았고, pore 갯수가 많이 없기 때문에
Second Run에서는 6조 시료 하나 만을 사용함.
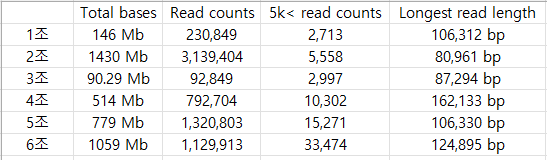
- Running을 할 때는 한 시료이지만, barcode 4개를 사용하여 되도록이면 많은 데이터를 얻고자 하였음.
- 그 결과 매우 정상적인 N50=17 kbp인 결과가 나옴. 즉, balanced sequencing option이 문제였다는 것이 확인됨.
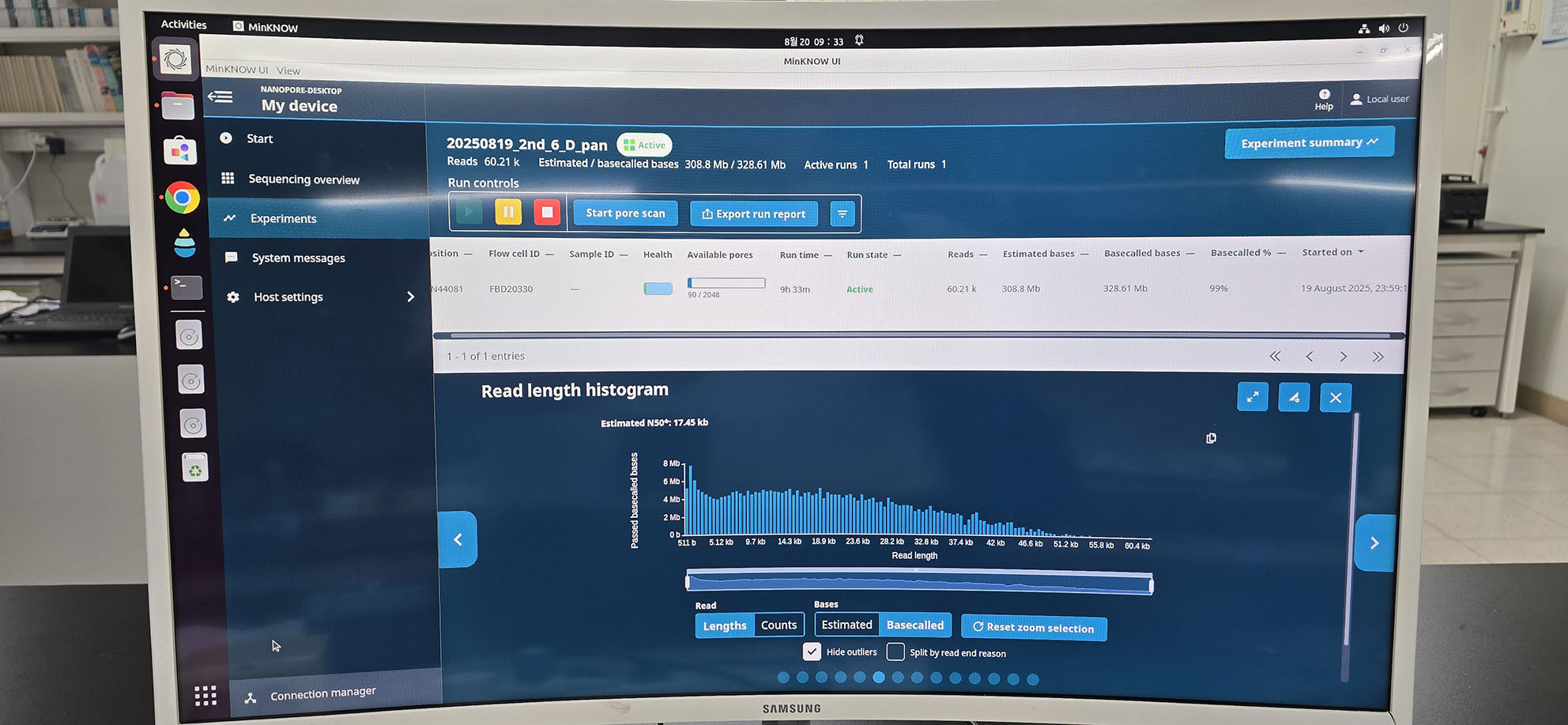
- 6조 시료를 약 0.4G 생성하였음.
3) Third RUN --- Balanced sequencing OFF, Old library kit 사용(6개월 정도 보관됨) (조교들이 수행함)
- Old library kit 의 이상유무를 확인하고자 balanced sequencing off 하여 같은 6조 시료를 사용하여 pore 100개인 flowcell로 실험함.
- 그 결과 N50=17 kbp 정도의 정상적인 결과가 나와서 old kit가 이상 없음이 확인됨.
- Third run은 100개의 pore만 남아있는 flowcell 이었지만, flowcell의 최대능력을 시험하고자 64 시간(2일 16시간)동안 실험한 결과,
총 1.26 Gbp의 데이터를 얻음. 특히, 1 Mbp 이상의 sequence도 확인됨.
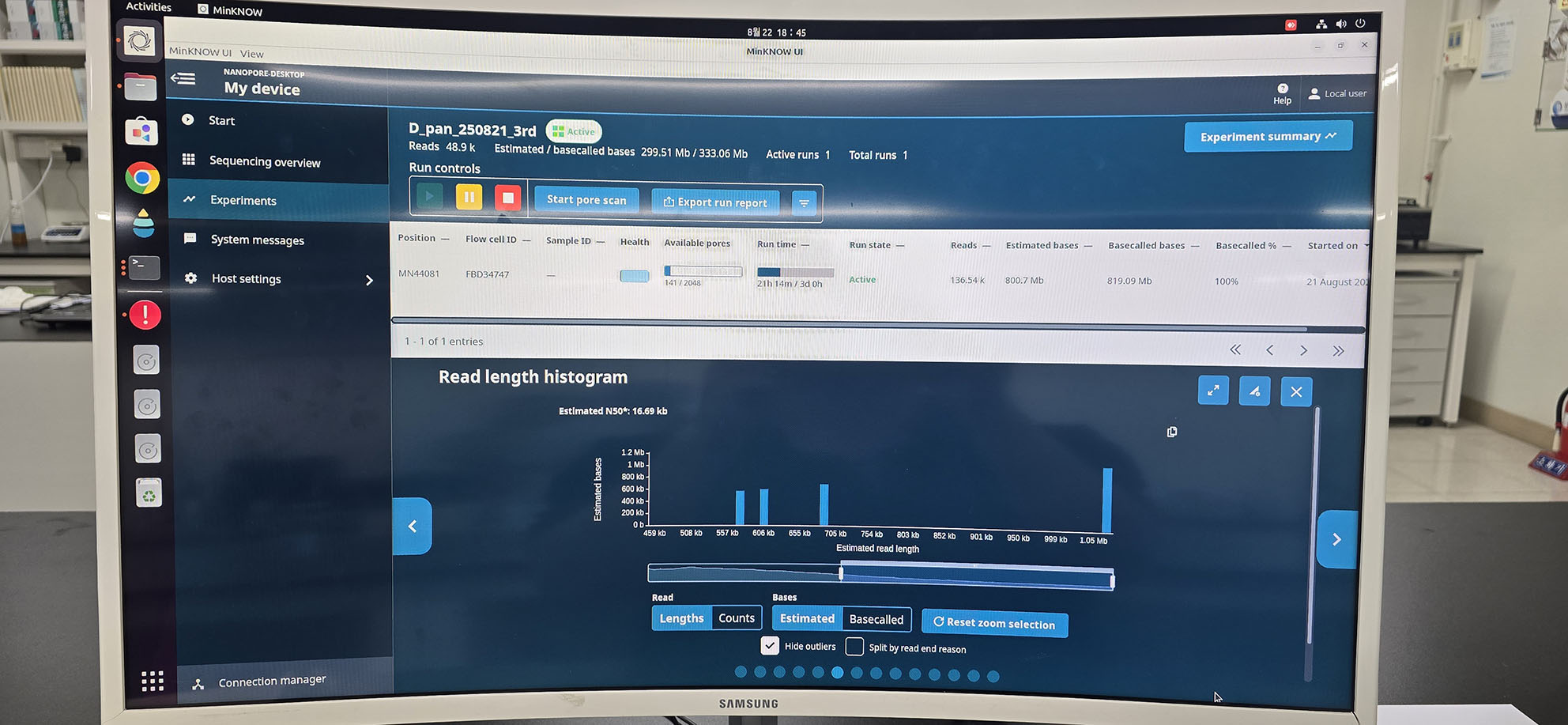
최종적으로 6조의 시료로 First, Second, and Third RUN을 합쳐서 약 2.5 Gbp 의 data를 얻었지만,
First RUN에서는 거의 모두가 짧은 서열의 data였음으로 실질적으로는 약 1.8 Gbp 의 data를 얻음.
이를 이용하여 assembly 과정을 진행함.
4) Fourth RUN --- Balanced sequencing OFF, New library kit 사용 (조교들이 수행함)
- ONT로부터 새 flowcell을 제공받아 DNA가 남아있는 1,2,5조 시료를 running 함.
125조 Nanopore report - 4th run
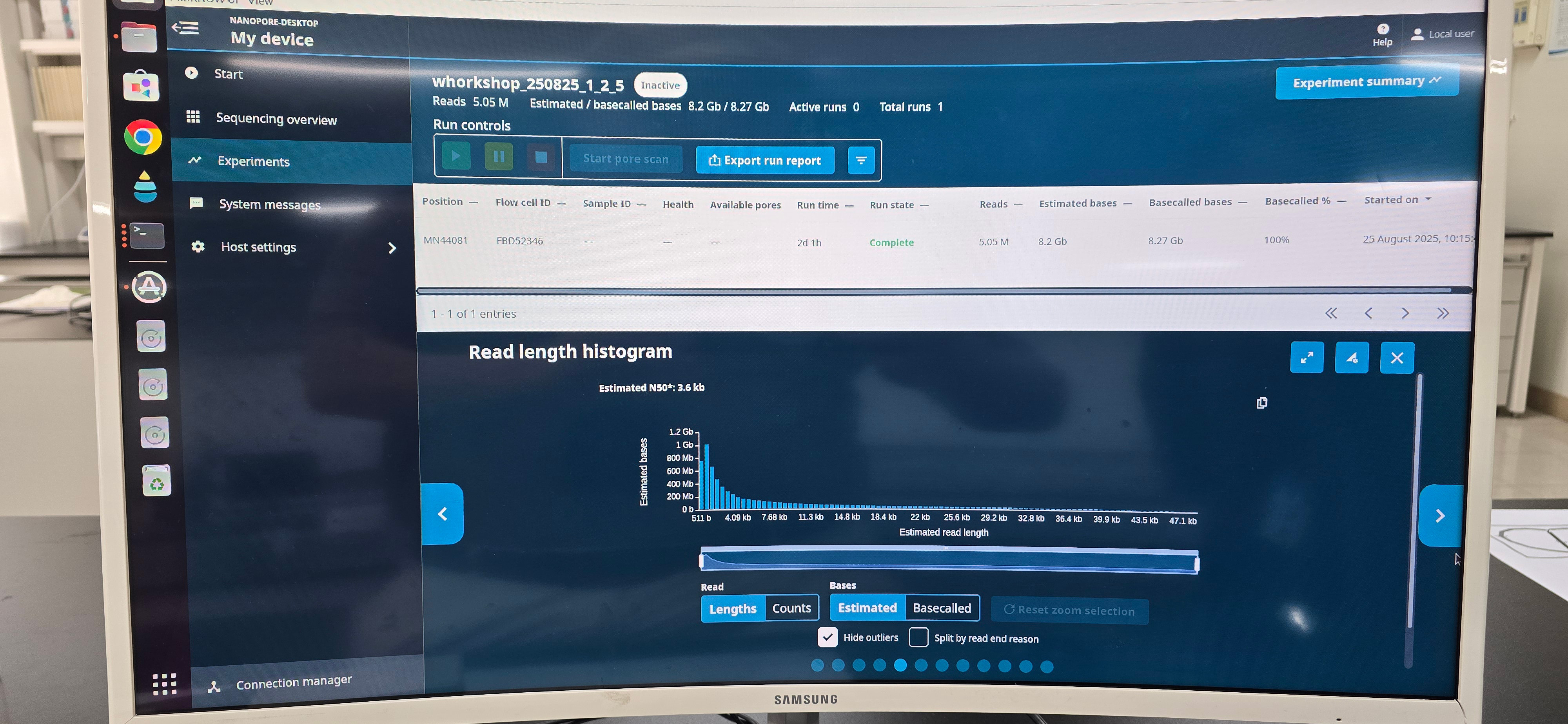
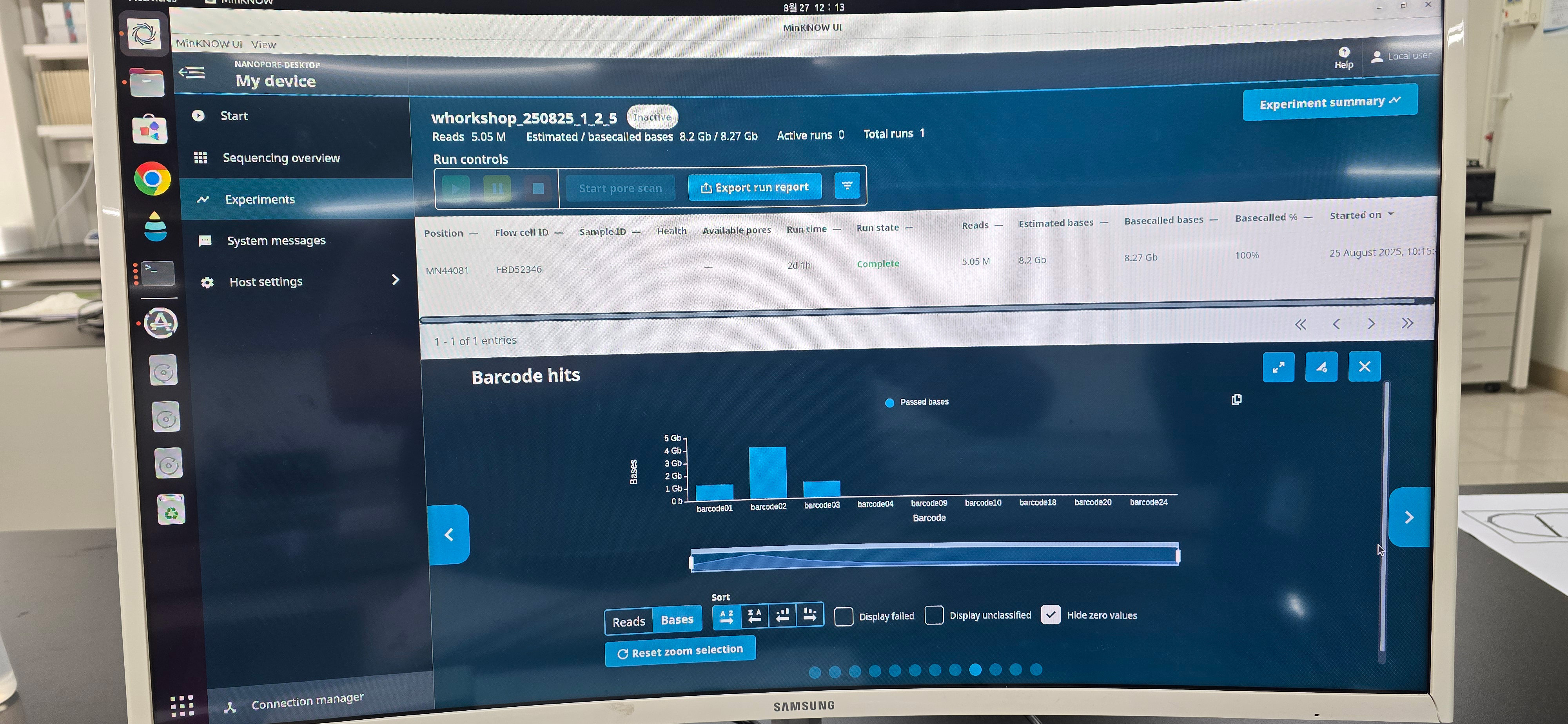
- 8.3 Gbp가 생성되었지만, 짧은 길이의 것이 많음. DNA가 추출 후 degradation 되었을 수도 있음.
- 이들 중 1조, 2조, 5조의 시료에서 동일한 데이터가 생성되지 못하고, 2조의 것이 4G 이상, 그리고 1조와 5조의 것은 각각 약 1 Gbp 만이 생산됨.
- Washing 후 pore의 갯수는 150개 정도밖에 남지 않아서 재실험 시 long-DNA를 생성하지 못한 3조와 4조 중 한 시료만을 시도하는 것이 바람직함.
MGI sequencing을 위한 QC 결과: 의뢰한 6개 시료 모두 통과함.
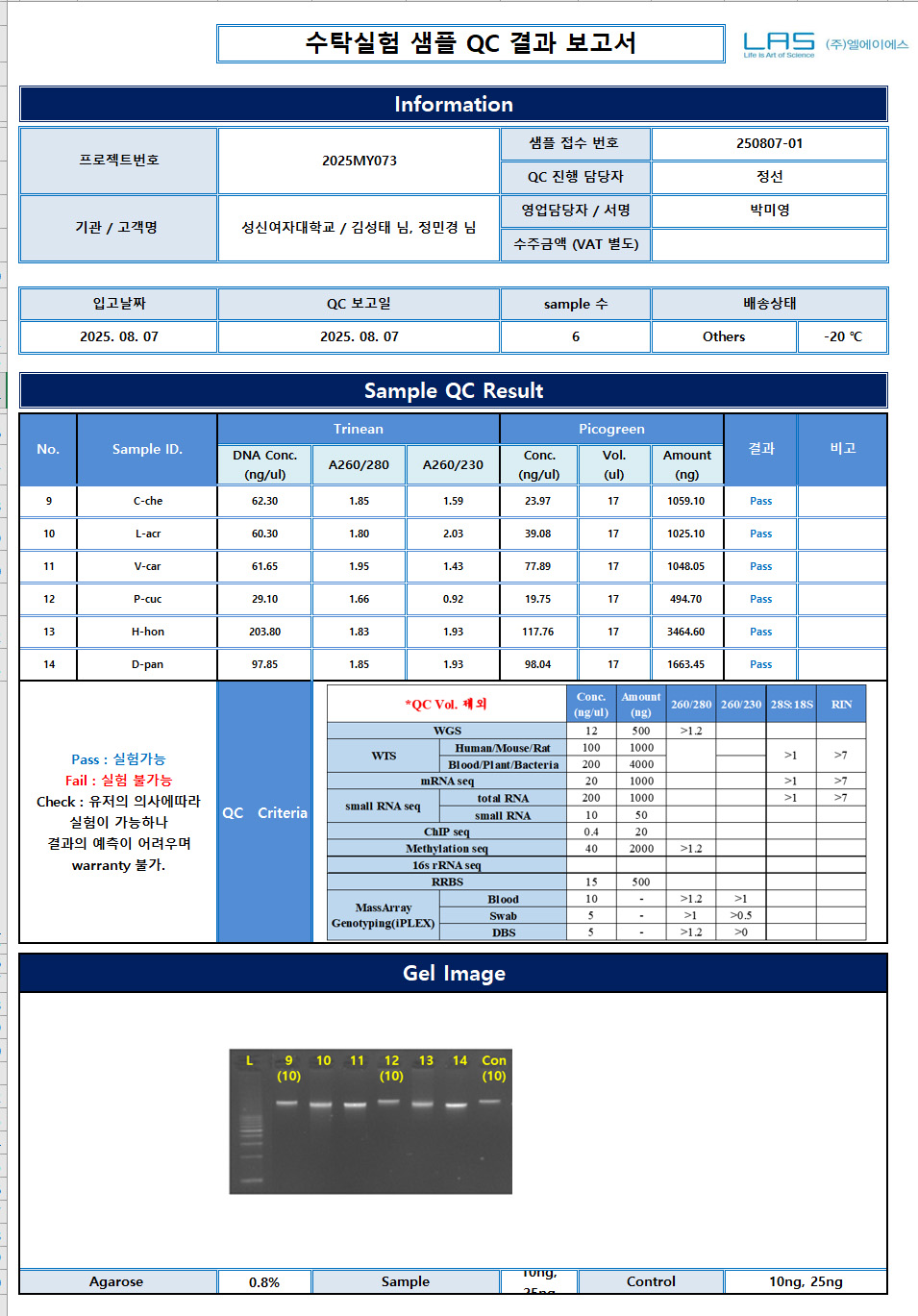
MGI sequencing 결과
6 종의 데이터에 대한 시퀀싱 결과 리포트 파일 다운로드
- 리포트에서 9~14번이 워크샵 결과임.
RESULT
I. MITOCHONDRIAL GENOME
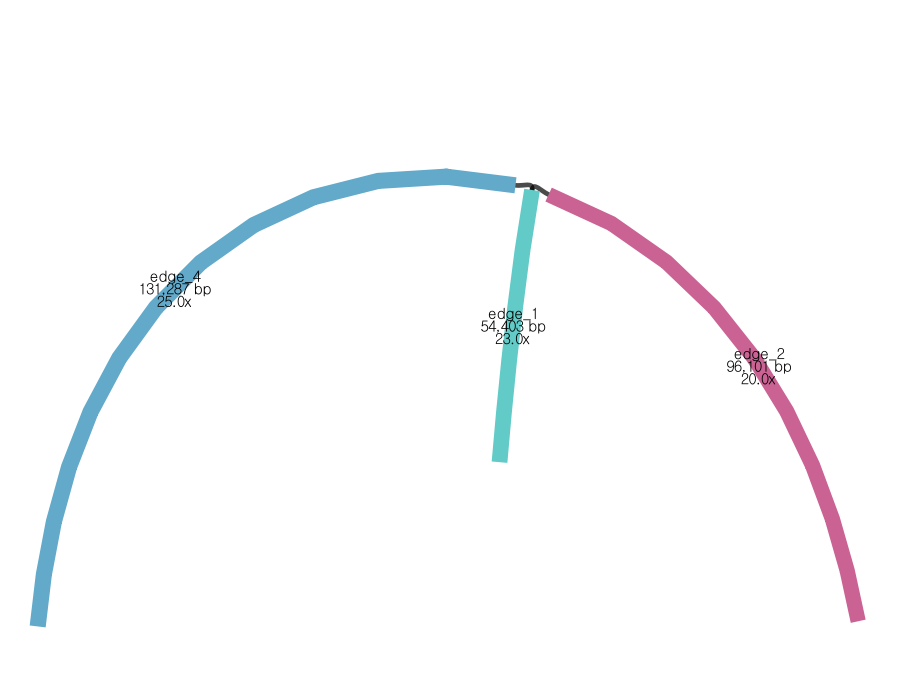
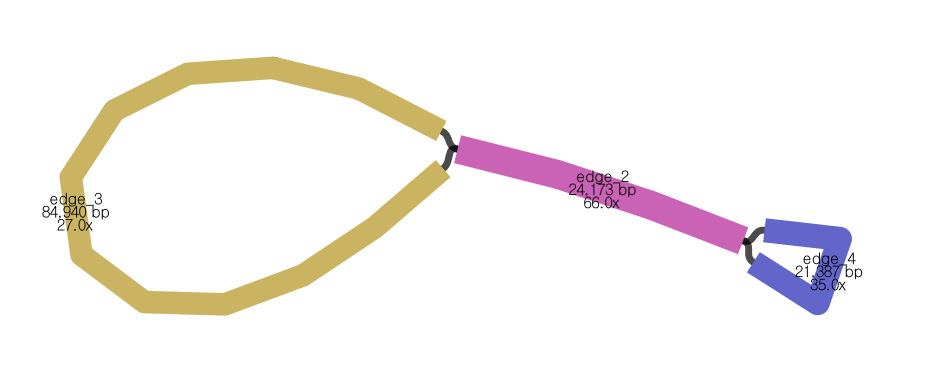
2조 MT genome of Lysimachia acroadenia: 281,790 bp
- 2조는 약 4 Gbp 정도의 long-read가 생성되었고, 8 kbp 이상의 long-read 만을 사용하여 assembly 한 결과 위와 같은 결과가 나왔음.
- 하나의 circular form으로 결과가 나오지는 않았고, 위 bandage graph는
1) egde4-edge1 (185,690 bp)과 2) edge2-edge1 (150,540 bp)의 두 가지 linear from으로 존재할 수 있음을 의미함.
- 세 edge를 합친 길이가 전체 genome 서열로 보고할 281,790 bp임.
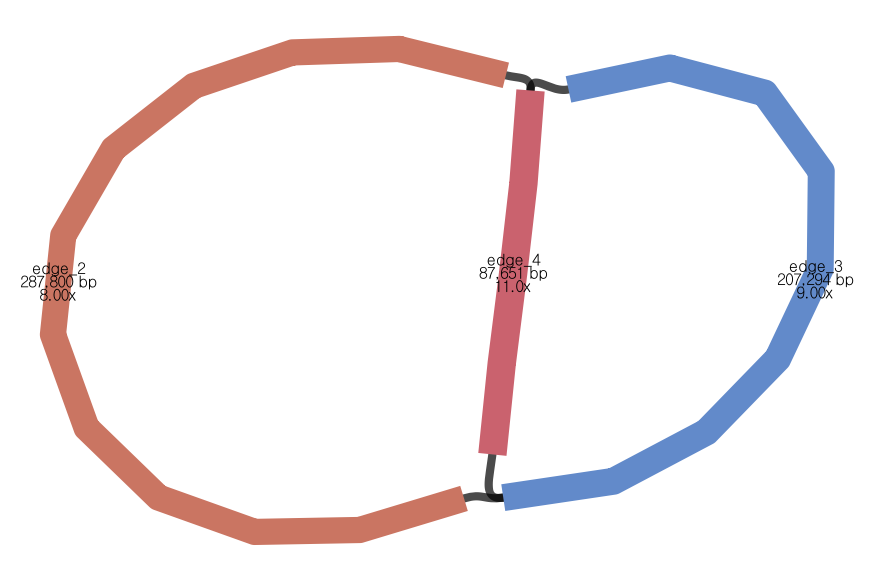
6조 MT genome of Deutzia paniculata: 582,750 bp
- 6조는 전체 2 Gbp 정도의 long-read가 생성되었는데, data양이 적어 coverage는 낮지만, report 할 수 있는 수준의 bandage graph가 형성되었습니다.
- 하나의 circular form으로 나오지는 않았으며, 위와 같이 bandage graph가 나왔다는 것은
1) edge3-edge4 의 subcircle과 2) edge2-edge3- edge4의 linear form 또는 edge2-edge4- edge3의 linear form이 존재할 수 있다는 의미입니다.
II. CHLOROPLAST GENOME
엽록체 유전체의 assembly는 POLAP에 의해 6종 모두 완벽하게 완성되었습니다.
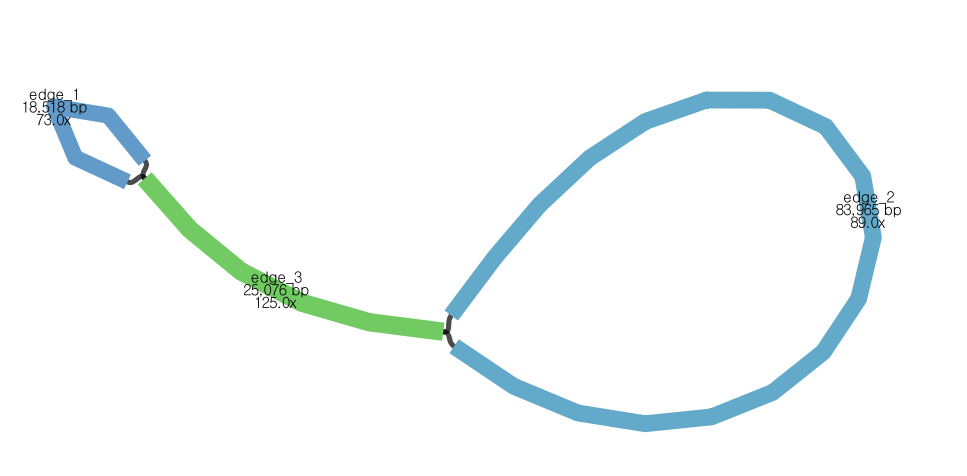
1조 CP genome of Crepidiastrum chelidoniifolium: 152,651 bp

2조 CP genome of Lysimachia acroadenia: 152,350 bp

3조 CP genome of Viburnum carlesii: 158,395 bp
(bandage에서는 IR이 인식되지 않았지만, annotation 해본 결과 IR이 인식되어 이상없이 나온 것 같습니다.)
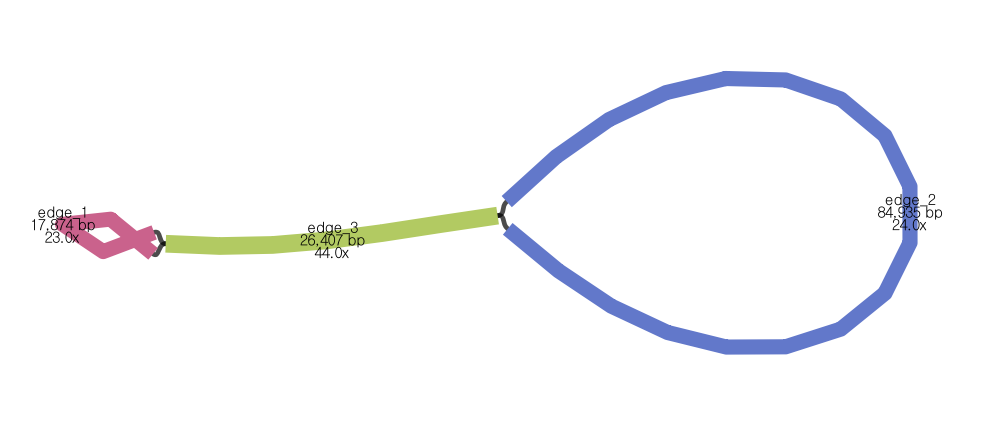
4조 CP genome of Ponerorchis cucullata: 155,658 bp
5조 CP genome of Hemerocallis hongdoensis: 150,742 bp

6조 CP genome of Deutzia paniculata: 157,068 bp
논문작성을 위한 임무

프로그램 수료식


=========================================================================================================================